中美贸易摩擦升级,国内企业如何破局?
2019年5月9日,美国政府宣布自2019年5月10日起,对从中国进口的2000亿美元清单商品加征的关税税率由10%提高到25%。5月13日,国务院关税税则委员会决定,自2019年6月1日起,对原产于美国的部分进口商品提高加征关税税率,其中诊断试剂(税号:30063000)属于加征25%关税税率的商品范围。
中美贸易摩擦持续升级,国内企业如何破局?2018年3月美国总统特朗普正式签署对华贸易备忘录,中美贸易战正式拉开帷幕。联川生物顺势而行,随即积极布局国内市场,战略性将美国LC Sciences国际研发中心基因捕获平台落地国内,打破基因捕获核心技术长久被外企垄断的现象,为国内企业提供高性价比一站式基因检测解决方案。
全球领先的寡核苷酸生产和供应商
美国LC Sciences(www.lcsciences.com)成立于2004年,联川生物国际研发中心,全球领先的寡核苷酸生产和供应商,全球5家拥有基因测序上游核心技术的公司之一,发明专利32项。公司生产的产品和服务遍及全球40多个国家,上万家单位和科研机构,依托自主μParaflo®微流体芯片合成寡核苷酸,推进基因捕获等领域的技术革新和发展。
自主技术包含μParaflo®微流体芯片平台,大规模寡核苷酸OligoMix®,一步式多重PCR解决方案VariantPro™,液相杂交捕获解决方案VariantBaits™等多种创新技术。

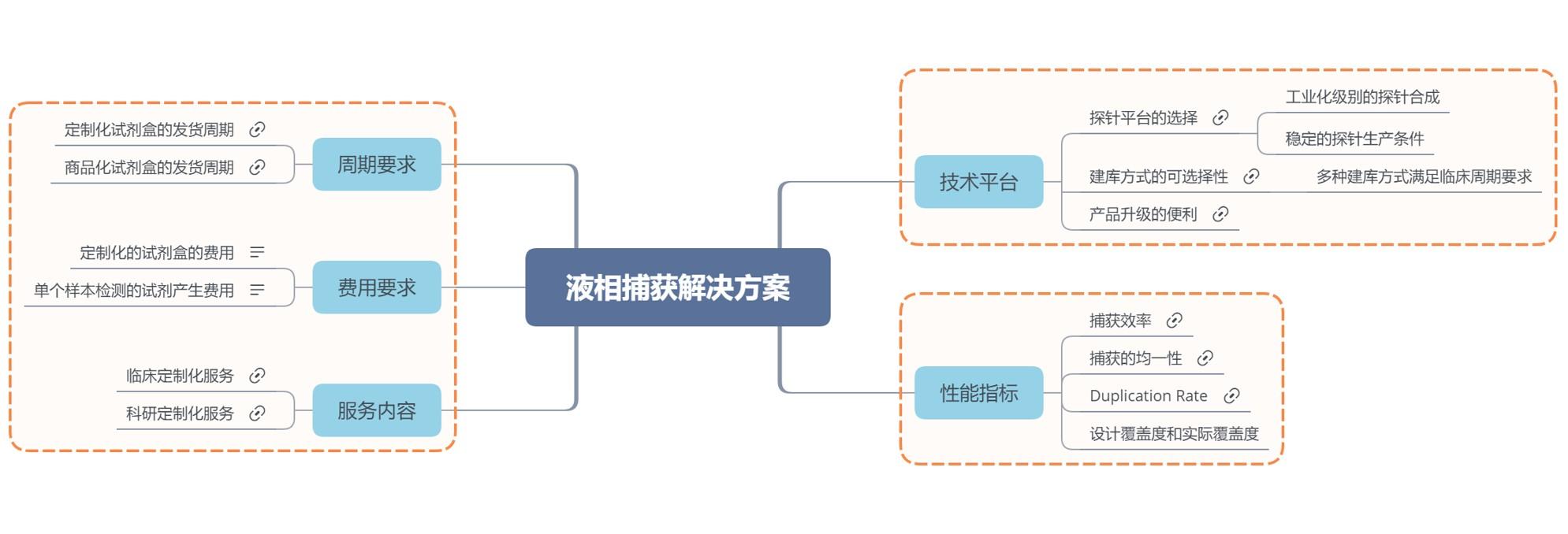
双V捕获平台临床NGS定制化试剂盒供应商
联川基因诊断创立于2016年,联川生物全资子公司,定位于体外诊断试剂盒开发与应用,将美国LC Sciences研发中心强大的双V捕获平台(VariantPro™&VariantBaits™)在国内GMP工厂落地生产,提供临床NGS定制化一站式检测解决方案。基于全球首创的VariantPro™一步法靶向基因捕获技术和自主知识产权的VariantBaits™液相杂交捕获系统,联川基因诊断专注为国内精准医学领域提供先进的基因检测解决方案和产品。
VariantBaits™液相杂交捕获系统
VariantBaits™液相杂交捕获系统可以对感兴趣的蛋白编码区域DNA或基因组上的特定序列进行富集,并在Illumina、Ion Torrent等二代测序平台进行高通量测序。通过自主专利的μParaflo®微流体芯片合成的高质量超长RNA探针,对带有测序接头的基因组文库进行液相杂交,与目标区域序列互补配对的探针在杂交时特异性结合目的片段DNA,通过链霉亲和素磁珠与探针上的生物素标记结合,从而抓取并富集目的片段DNA。
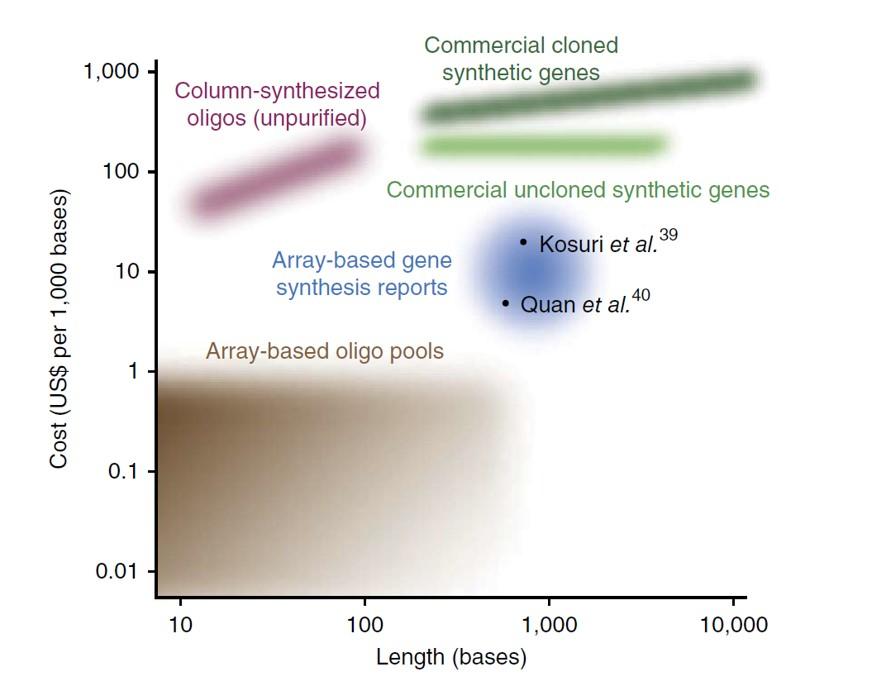
μParaflo® Microfluidics工业级核酸合成解决方案

根植于光原位合成原理,联川研发出以硅晶板为基础的大规模合成芯片平台μParaflo® Microfluidics,是目前少数几家拥有此项技术的公司。通过微流控技术对核酸的原料进行控制,经由控制光的封闭与否对合成的DNA反应进行控制,达到合成DNA的目的。相比于其他方式,此种DNA合成可以实现更大规模的合成以及更可控的DNA合成质量。
在合成DNA时,当长度小于1000bp时,芯片合成成本远低于其他合成方式。以此表明,在大规模合成DNA的领域当中,芯片合成占有绝对优势,也是主流选择。
Kosuri, S., & Church,G. M. (2014). Large-scale de novo DNA synthesis: technologies and applications.Nature methods, 11(5), 499.
μParaflo® Microfluidics芯片平台可一次性合成30,000根寡核苷酸,可实现大规模探针合成,更适用于二代测序靶向捕获。μParaflo® Microfluidics平台自2004年以来发表数十篇文章,其中多篇发表在CNS顶级杂志上。(文章见文末列表)
VariantBaits™探针优势

结合力优势:RNA/DNA>DNA/DNA
RNA探针对基因组文库(DNA)结合力更强,这使得RNA探针相比于DNA探针具有更低的退火温度,实验中更好的稳定性。
设计优势:覆瓦式和长探针
探针长度和探针设计排布的策略对捕获效率有更多的影响,覆瓦式(Tiling)的探针设计比相邻式或间隔式的探针排布富集效率更高;长探针在捕获时对目标序列的错配容忍度更高,因此长探针在捕获indels上灵敏度更高。
Clark, Michael J., et al."Performance comparison of exome DNA sequencing technologies." Naturebiotechnology 29.10 (2011): 908.3
VariantBaits™技术优势
国内唯一一家大规模自主核心技术的探针合成平台,探针可靠性已获数十项应用验证
120nt超长RNA捕获探针覆瓦式覆盖目标区域,拥有更高的错配容忍度和捕获效率
独特的探针设计优化,充分利用探针与捕获区域的结合特性,减少冗余产生
对于高GC区域,进行独特的探针方法设计,最大限度上捕获到高GC区域
可按客户需求个性化定制Panel,Panel定制30个工作日,可同时定制多种Panel
VariantBaits™性能展示
VariantBaits™良好的捕获效率
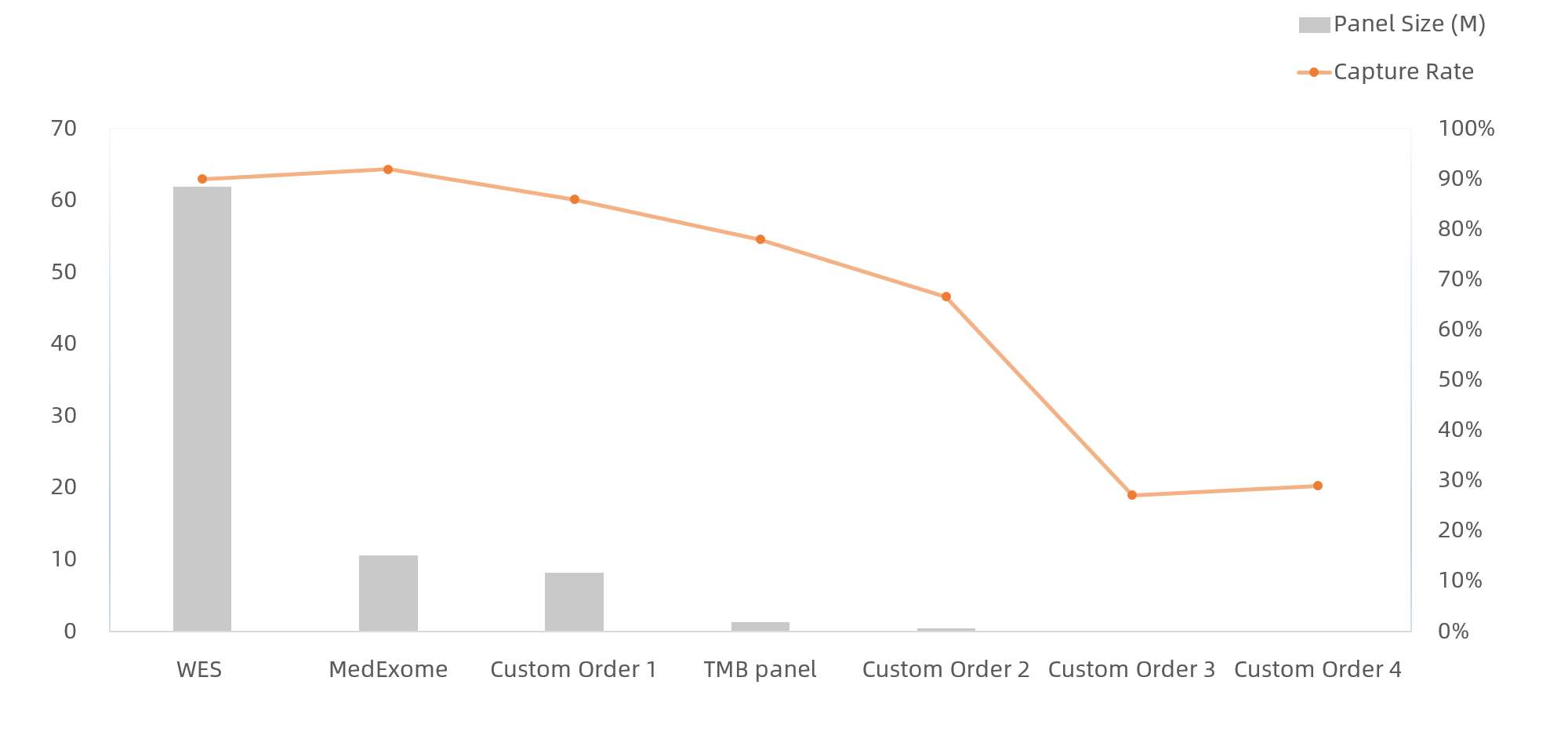 VariantBaits™优异的覆盖度
VariantBaits™优异的覆盖度
 VariantBaits™均一化的覆盖度
VariantBaits™均一化的覆盖度
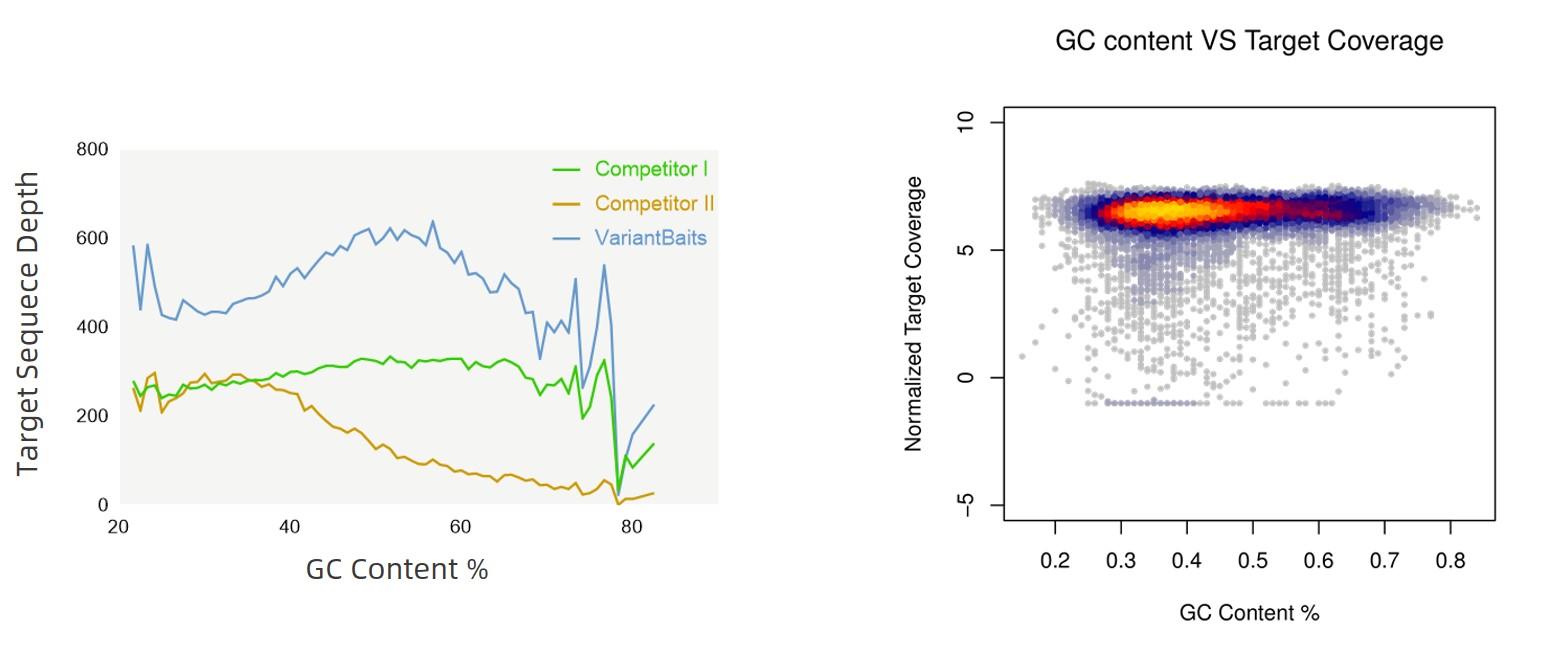
VariantBaits™稳定的重复表现
 VariantBaits™产品性能
VariantBaits™产品性能
 VariantBaits™建库原理
VariantBaits™建库原理
 VariantBaits™定制流程
VariantBaits™定制流程
 VariantBaits™部分合作成果
1.Gao X, Church, GM, et al. (2004) Accurate multiplex gene synthesis from programmable DNA chips. Nature 432, 1050-1054.
2.Porreca GJ, Zhang K, Li JB, Xie B, Austin D, Vassallo SL, LeProust EM, Peck BJ, Emig CJ, Dahl F, Gao Y, Church GM, Shendure J. (2007) Multiplex amplification of large sets of human exons. Nat Methods 4(11), 931-36.
3.Gnirke A, Melnikov A, Maguire J, et al. (2009) Solution hybrid selection with ultra-long oligonucleotides for massively parallel targeted sequencing. Nat Biotechnol 27, 182–89.
4.Lira Mamanova, Alison J Coffey1, et al. (2010) Target-enrichment strategies for nextgeneration sequencing. Nat Methods 7(2):111-8.
5.Teer JK, Bonnycastle LL, et al. (2010) Systematic comparison of three genomic enrichment methods for massively parallel DNA sequencing. Genome Res 20(10), 1420-31.
6.Matzas M, St?hler PF, et al. (2010) High-fidelity gene synthesis by retrieval of sequence-verified DNA identified using high-throughput pyrosequencing. Nat Biotechnol 28(12), 1291-94.
7.Nautiyal S, Carlton VE, Lu Y, Ireland JS, Flaucher D, Moorhead M, Gray JW, Spellman P, Mindrinos M, Berg P, Faham M. (2010) High-throughput method for analyzing methylation of CpGs in targeted genomic regions. Proc Natl Acad Sci 107(28), 12587-92.
8.Myllykangas S, Buenrostro JD, et al. (2011) Efficient targeted resequencing of human germline and cancer genomes by oligonucleotide-selective sequencing. Nat Biotechnol 29, 1024-27.
9.Diep D, Plongthongkum N, Gore A, Fung H, Shoemaker R, Zhang K. (2012) Library-free methylation sequencing with bisulfite padlock probes. Nature Methods 9(3), 270-2.
10.Labrie V, Buske OJ, Oh E, Jeremian R, Ptak C, Gasiūnas G, Maleckas A, Petereit R, ?virbliene A, Adamonis K et al.(2016) Lactase nonpersistence is directed by DNA-variation-dependent epigenetic aging. Nature Structural &Molecular Biology 23(6):566-73.
11.Zaccai F, C T, Savitzki D, Zivony-Elboum Y, Vilboux T, Fitts EC, Shoval Y, Kalfon L, Samra N, Keren Z. (2017) Phospholipase A2-activating protein is associated with a novel form of leukoencephalopathy. Brain 140(2), 370-386.
VariantBaits™部分合作成果
1.Gao X, Church, GM, et al. (2004) Accurate multiplex gene synthesis from programmable DNA chips. Nature 432, 1050-1054.
2.Porreca GJ, Zhang K, Li JB, Xie B, Austin D, Vassallo SL, LeProust EM, Peck BJ, Emig CJ, Dahl F, Gao Y, Church GM, Shendure J. (2007) Multiplex amplification of large sets of human exons. Nat Methods 4(11), 931-36.
3.Gnirke A, Melnikov A, Maguire J, et al. (2009) Solution hybrid selection with ultra-long oligonucleotides for massively parallel targeted sequencing. Nat Biotechnol 27, 182–89.
4.Lira Mamanova, Alison J Coffey1, et al. (2010) Target-enrichment strategies for nextgeneration sequencing. Nat Methods 7(2):111-8.
5.Teer JK, Bonnycastle LL, et al. (2010) Systematic comparison of three genomic enrichment methods for massively parallel DNA sequencing. Genome Res 20(10), 1420-31.
6.Matzas M, St?hler PF, et al. (2010) High-fidelity gene synthesis by retrieval of sequence-verified DNA identified using high-throughput pyrosequencing. Nat Biotechnol 28(12), 1291-94.
7.Nautiyal S, Carlton VE, Lu Y, Ireland JS, Flaucher D, Moorhead M, Gray JW, Spellman P, Mindrinos M, Berg P, Faham M. (2010) High-throughput method for analyzing methylation of CpGs in targeted genomic regions. Proc Natl Acad Sci 107(28), 12587-92.
8.Myllykangas S, Buenrostro JD, et al. (2011) Efficient targeted resequencing of human germline and cancer genomes by oligonucleotide-selective sequencing. Nat Biotechnol 29, 1024-27.
9.Diep D, Plongthongkum N, Gore A, Fung H, Shoemaker R, Zhang K. (2012) Library-free methylation sequencing with bisulfite padlock probes. Nature Methods 9(3), 270-2.
10.Labrie V, Buske OJ, Oh E, Jeremian R, Ptak C, Gasiūnas G, Maleckas A, Petereit R, ?virbliene A, Adamonis K et al.(2016) Lactase nonpersistence is directed by DNA-variation-dependent epigenetic aging. Nature Structural &Molecular Biology 23(6):566-73.
11.Zaccai F, C T, Savitzki D, Zivony-Elboum Y, Vilboux T, Fitts EC, Shoval Y, Kalfon L, Samra N, Keren Z. (2017) Phospholipase A2-activating protein is associated with a novel form of leukoencephalopathy. Brain 140(2), 370-386.