基因组重测序是对已知基因组序列的物种进行全基因组范围的测序,并在此基础上对个体或群体进行差异性(SNP、InDel和SV等)分析。基于基因组重测序技术,可以快速进行资源普查筛选,寻找到大量遗传变异,实现遗传进化分析及重要性状候选基因的预测。随着测序成本降低以及拥有参考基因组序列物种增多,基因组重测序成为研究遗传育种和群体进化的有效方法。
在全基因组水平检测与表型关联的高频、低频、甚至是罕见的点突变及结构变异信息
对于个体样本,可获得全面的基因组突变谱信息
对于群体样本,可进一步研究物种的进化历史、环境适应性、自然选择和性状定位
技术路线

细胞,组织,全血,总DNA等
建议总DNA起始量:5 μg,最低0.5 μg,浓度≥300 ng/μL(Qubit定量)
A:重测序一般可以检测单核苷酸多态性(SNP)、插入缺失(InDel)、拷贝数变异(CNV)、染色体结构变异(SV)等。
A:测序深度根据研究目的、样本量及合作伙伴的预期而定。30×测序深度即可检测绝大部分SNV,但如果客户的研究目的是寻找癌组织中较大的结构变异、少数肿瘤细胞携带的丰度较低的突变,建议测序深度(一般)至少50×以上;群体重测序可以使用较低深度测序(~10×),用群体分析策略寻找相关变异。
A:FFPE样本提取的DNA多数存在降解的情况,基因组呈现片段化,CNV/SV等结构性变异检出的假阳性率较高,无法体现全基因组测序在结构变异检测方面的优势,且通过增加测序深度提高变异检出准确性的成本太高。
A:全外显子组测序捕获基因组的外显子区域,其基因组信息约占基因组大小的1.5%;全基因组测序对于全基因组层面来说,变异信息更全面,没有止步于编码区,而是向整个非编码区扩展,性价比更高,平均数据单价较全外显子组测序便宜了5倍以上。近些年非编码区突变的研究越来越多,其可与多种癌症在内的复杂疾病发生相关。
全基因组测序鉴定卵巢癌化疗抗性特征
研究背景
过去的30年间,高级别浆液性卵巢癌(High-grade serous ovarian cancer, HGSC)患者的存活情况几乎没有改善,标准治疗的方法未见比以铂类为主要成分的联合化疗方法效果有提高。本文研究化疗选择下HGSC基因组进化,以期发现如何克服化疗抗性产生的治疗方法。
方法流程
取材:92个HGSC患者原发实体瘤、腹水样本或尸检样本共114个样本及正常对照
测序: 1. HiSeq 2000,PE 100 bp ; 2. WGS:Tumor 52×、Normal 40× ; 3. 转录、甲基化、miRNA支撑WGS数据
分析: 1. 与参考基因组GRCh37进行比对 2. 检测somatic mutation 3. 高频突变基因筛选 4. 突变特征分析 5. 融合基因检测 6. 分子分型
80个实体瘤和12个腹水样本共找到36,561个SVs,其中每个样本SV数目在48~1,064个,所有样本TP53突变普遍存在。另有超过一半的原发瘤样本,同源重组修复功能受损或者BRCA甲基化。多个样本抑癌基因RB1、NF1、RAD51B、PTEN 和化疗抗性基因CCNE1 发生突变,且CCNE1 基因突变与同源重组通路不同。
发现样本以BRCA signatrue和Age signatrue为主。如果样本只是产生BRCA 基因突变,那么进行化疗是敏感的,但一旦样本出现CCNE1 基因突变,那么个体进行化疗比较难治疗或者会产生抗性。并且结合临床研究,出现CCNE1 突变的患者预后比较差。
患者化疗后其编码区和非编码区SNV/InDel数量增多,且化疗后产生治疗抗性的患者存在BRCA1启动子去甲基化、BRCA1 / BRCA2 功能恢复性突变、SLC25A40-ABCB1基因融合的情况。
研究结论
通过对HGSC患者的成对样本进行WGS研究,重点关注前期化疗有效且后期化疗产生抗性的患病个体。在产生化疗抗性的个体中频繁检测到CCNE1基因突变,说明CCNE1基因突变意味着预后差。通过检测不同治疗阶段癌细胞的变异情况,发现基因断裂导致抑癌基因失活是产生化疗抗性的重要原因。此外还观测到一些分子事件与获得性抗性相关,如BRCA1或BRCA2 reversion,BRCA1启动子去甲基化,周期性启动子融合。此项研究揭示了在化疗选择压力下HGSC患者基因组的异质性和适应性,在选择化疗方案作为HGSC治疗手段时,需要采用必要的策略避免产生化疗抗性。
参考文献
Patch A M, Christie E L, Etemadmoghadam D, et al. Whole–genome characterization of chemoresistant ovarian cancer[J]. Nature, 2015, 521(7553):489-94.
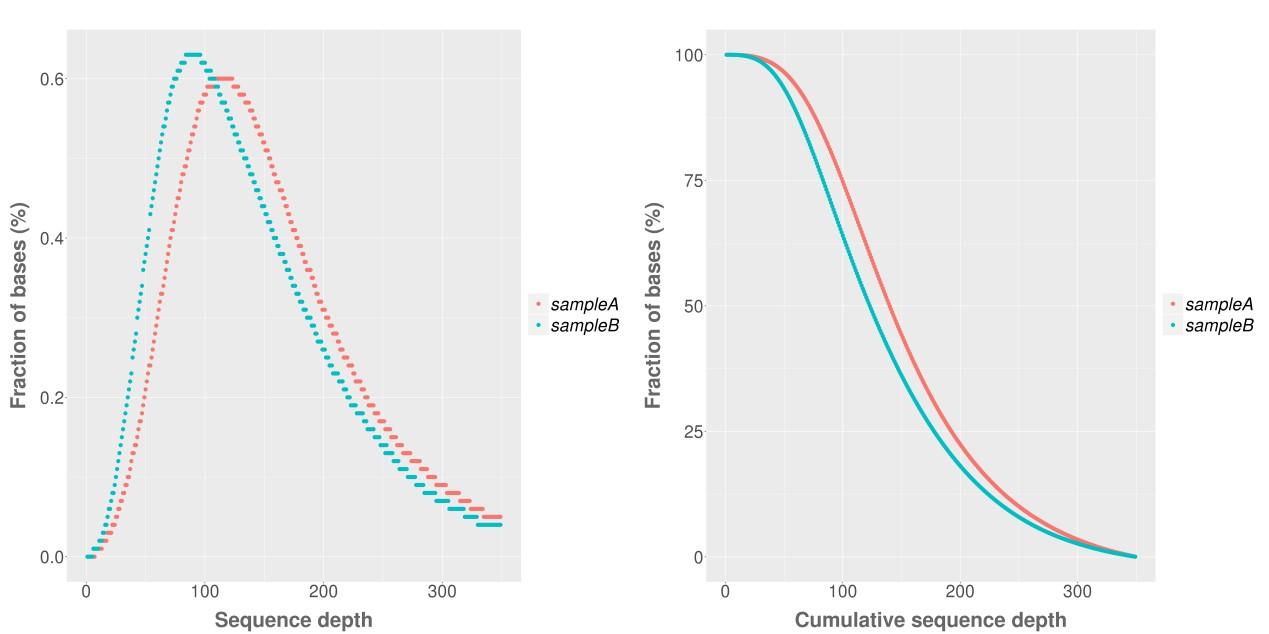
测序深度图利用重复标记后的比对结果进行覆盖度,深度等的统计。左图为不同 的测序深度的碱基比例。横坐标表示测序深度,纵坐标表示测序深度为x的碱基在所有碱基中的比例;右图为不同测序深度上的累积碱基比例,横 坐标表示测序深度,纵坐标表示测序深度超过x的碱基在所有碱基中的比例。 比如测序深度为50X对应的碱基比例约为95%,表示约有95%的碱基其测 序深度大于50X。
染色体覆盖深度图横坐标表示各染色体,纵坐标表示平均覆盖深度。

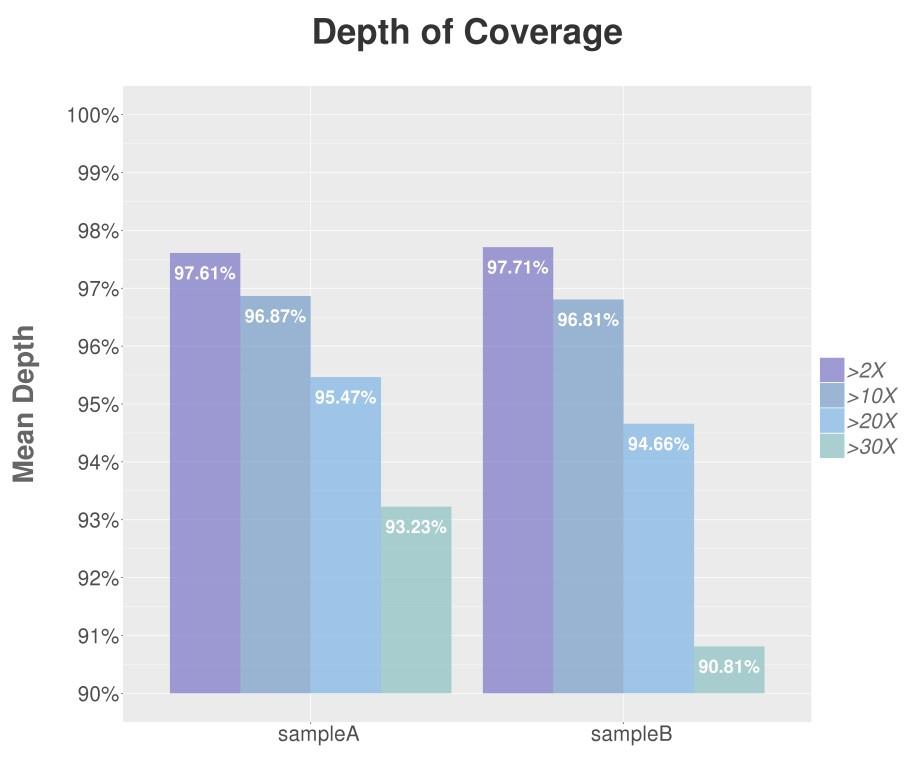
Reads覆盖度阶梯图纵坐标为测序深度对应的Reads的覆盖比例,不同颜色代表不同的测序深度,图中 样本A 93.23%的Reads覆盖了20X以上,样本B 94.66%的Reads覆盖了20X以上。
基因组和外显子区域SNP特征图第1个柱状图中,Hom代表纯合,其纵坐标代表基因型为纯合的SNP数目;Het代表杂合,其纵坐标代表基因型为杂合的SNP数目, Het rate代表杂合基因型的SNP在所有SNP中的比例。第2个柱状图中,tv代表颠换,其纵坐标代表tv的SNP数目;ts代表转换,其纵坐标代表ts的SNP数目。ts/tv是转换/颠换的比值。

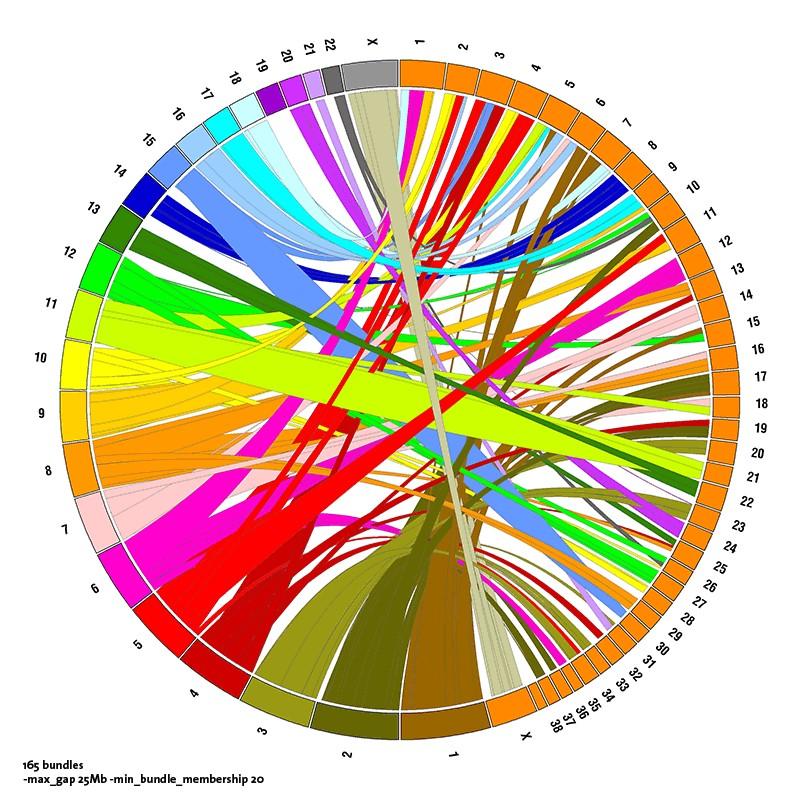
SV circos图选取测序质量值top20个SV进行了圈图绘制,圈图中间弧形连接的片段发生了染色体结构变异,位置发生了重排。